発表のポイント
- 東北大学大学院生命科学研究科・近藤倫生教授らの提案「”Holistic Genomic Approach to Asia-Pacific Marine Biodiversity”(邦題:アジア太平洋生物多様性への全ゲノムアプローチ」が公益財団法人笹川平和財団海洋政策研究所のOcean shotに採択されました。
- 環境DNA情報を用いて、海洋における新種の発見、種の分布境界の特定など、海洋生物多様性の理解を大きく前進させることが期待されます。
概要
東北大学大学院生命科学研究科の近藤倫生教授と広島修道大学人間環境学部の岡西政典助教らの共同研究グループの研究課題が、笹川平和財団海洋政策研究所のOcean Shotに採択されました。Ocean Shotは、ムーンショット型研究開発制度の海洋版として設立され、新たな海洋の生物種や生態、その機能の発見、海洋での発見を支援する新しいデータや技術の発見のための研究を大規模に支援するプログラムです。
本研究課題では、海洋生態系の科学的な理解とその持続的な利用の基礎的手法を構築することを目指し、日本近海の海洋生物を網羅するDNA情報を収集し、生物多様性評価を行う上での基盤を形成します。また、日本から太平洋における海洋生物の空間分布を,環境DNA分析手法によって高解像度で明らかにします。広範な汎太平洋環境で得られたDNA調査の結果は、専用データベース "ANEMONE DB "として公開します。
これらを統合的に解析することで、「生物多様性のダークマター」と呼ばれる、謎の多い海洋生物の生物多様性について、そのホットスポットや分布の境界など、のグローバルな海洋生物の分布パターンを明らかにできることが期待できます。
本研究課題では、海洋生態系の科学的な理解とその持続的な利用の基礎的手法を構築することを目指し、日本近海の海洋生物を網羅するDNA情報を収集し、生物多様性評価を行う上での基盤を形成します。また、日本から太平洋における海洋生物の空間分布を,環境DNA分析手法によって高解像度で明らかにします。広範な汎太平洋環境で得られたDNA調査の結果は、専用データベース "ANEMONE DB "として公開します。
これらを統合的に解析することで、「生物多様性のダークマター」と呼ばれる、謎の多い海洋生物の生物多様性について、そのホットスポットや分布の境界など、のグローバルな海洋生物の分布パターンを明らかにできることが期待できます。
詳細の説明
研究の背景
生態系において、どこにどのような生物がどれくらい存在しているのかという課題は、生態系の科学的理解とその持続可能な利用にとって極めて重要です。しかし生態学においてこの課題は長年議論されており、特に地球規模での解明はなされていません。
その第一の理由は、「生物多様性のダークマター」と呼ばれる、未報告の海洋生物の新種の存在です。推定200万種とも言われる海洋生物の新種が推定される背景には、形態だけでは区別できない「隠蔽種」の存在が挙げられます。この問題を解決するためには、見過ごされてきた隠蔽種を遺伝子レベルで区別・発見し、新種として記載(注1)することが不可欠です。
第二の理由は、海洋生物の分布調査が困難な点です。特に海洋面積の90%以上を占める深海域(水深200 m超)では、包括的な生物調査自体にコストがかかり、生物多様性のホットスポットや分布の境界はどこにあるのか?それは分類群によって異なるのか?などの基本的な知見も明らかになっておらず、定量的な評価方法の確立を妨げています。
この問題を、近年のDNA解析技術の発展が解決しつつあります。環境DNAメタバーコーディングやゲノムワイド系統解析と呼ばれる技術の進歩です。この方法により、生物種数の解明や、その定量評価の効率、ならびに隠蔽種の検出に不可欠な、大規模なサンプルに基づく遺伝的多様性の解析効率は大幅に向上しました。しかしその基盤となる網羅的なDNAバーコードライブラリー(注2)や、観測基盤が十分とは言えず、生物多様性の全球的な把握には至っていません。
そのため遺伝子解析による隠蔽種の判定やDNAバーコードライブラリーの整備、広範な観測基盤の整備は、生物多様性の分布を網羅的に把握するために不可欠なステップです。これらが整備されることで、海洋生物のダークマターの存在量が明らかになり、海洋生態系の保全や持続可能な管理のための重要な知見をもたらされると期待できます。
その第一の理由は、「生物多様性のダークマター」と呼ばれる、未報告の海洋生物の新種の存在です。推定200万種とも言われる海洋生物の新種が推定される背景には、形態だけでは区別できない「隠蔽種」の存在が挙げられます。この問題を解決するためには、見過ごされてきた隠蔽種を遺伝子レベルで区別・発見し、新種として記載(注1)することが不可欠です。
第二の理由は、海洋生物の分布調査が困難な点です。特に海洋面積の90%以上を占める深海域(水深200 m超)では、包括的な生物調査自体にコストがかかり、生物多様性のホットスポットや分布の境界はどこにあるのか?それは分類群によって異なるのか?などの基本的な知見も明らかになっておらず、定量的な評価方法の確立を妨げています。
この問題を、近年のDNA解析技術の発展が解決しつつあります。環境DNAメタバーコーディングやゲノムワイド系統解析と呼ばれる技術の進歩です。この方法により、生物種数の解明や、その定量評価の効率、ならびに隠蔽種の検出に不可欠な、大規模なサンプルに基づく遺伝的多様性の解析効率は大幅に向上しました。しかしその基盤となる網羅的なDNAバーコードライブラリー(注2)や、観測基盤が十分とは言えず、生物多様性の全球的な把握には至っていません。
そのため遺伝子解析による隠蔽種の判定やDNAバーコードライブラリーの整備、広範な観測基盤の整備は、生物多様性の分布を網羅的に把握するために不可欠なステップです。これらが整備されることで、海洋生物のダークマターの存在量が明らかになり、海洋生態系の保全や持続可能な管理のための重要な知見をもたらされると期待できます。
今回の取り組み
このような背景を受け本研究課題では、環境DNA解析とゲノムワイドDNA解析手法を用いて、これまで不可能であった海洋全体の主要な生物群を網羅した生物多様性の定量的評価と分布推定を目指します。本プロジェクト代表者である近藤博士が運営する環境DNAを活用した生物多様性観測プラットフォーム「ANEMONE(All Nippon eDNA Monitoring Network)」と(図1)、東北大学の須山博士が開発した、様々な生物種の遺伝的多様性データを効率的かつ迅速に取得できる「MIG-seq(multiplexed ISSR genotyping by sequencing)法」を用い、以下の主要な目標を設定します:
1. 隠蔽種、新種の発見:海洋に広く見られる甲殻類(エビ・カニ・ヤドカリなど)、海綿動物(注3)、刺胞動物(イソギンチャク・サンゴなど)、軟体動物(主に巻貝、二枚貝、イカ・タコ)、環形動物(主にゴカイ)、棘皮動物(ウニ・ヒトデ・ナマコなど)や、体長1 mm以下の微小なメイオベントスの専門家が収集した20万超の標本からDNAを抽出し、ゲノムワイドDNA解析を行います(図2)。これにより、その遺伝的分化、多様性分布、隠蔽(亜)種の検出を行い、従来の形態に基づくアプローチよりも効率的な新種発見手法を確立します。
2. DNAバーコードライブラリーの構築:上記の解析で得られるDNA配列を蓄積し、日本近海の海洋生物との種内変異を網羅するDNAバーコードライブラリーを完成させ、オープンデータベースとして公開します。
3. 生物多様性の分布パターンの解明:汎太平洋地域における各国(ベトナム、タイ、マレーシア、シンガポール、インドネシア、フィリピン、韓国、オーストラリア、ニュージーランド)の新規環境DNAサンプルと、日本郵船との共同研究から得られる太平洋横断型の外洋の環境DNAサンプルを解析します。得られたデータから、海洋生物の組成変化を示す生物多様性ホットスポットや海洋生物多様性のグローバルなパターンを解明します。これらの調査は、ANEMONE調査プロトコルに基づき,環境DNA学会によって確立された標準化された方法を利用し、後世の研究との比較ができるように取り組みます。
本研究開発への参加・協力機関(五十音順):
石巻専修大学、かずさDNA研究所、熊本大学、島根大学、筑波大学、東京大学、東北大学、日本郵船、八戸工業大学、広島修道大学、北海道大学、和歌山高専、琉球大学
1. 隠蔽種、新種の発見:海洋に広く見られる甲殻類(エビ・カニ・ヤドカリなど)、海綿動物(注3)、刺胞動物(イソギンチャク・サンゴなど)、軟体動物(主に巻貝、二枚貝、イカ・タコ)、環形動物(主にゴカイ)、棘皮動物(ウニ・ヒトデ・ナマコなど)や、体長1 mm以下の微小なメイオベントスの専門家が収集した20万超の標本からDNAを抽出し、ゲノムワイドDNA解析を行います(図2)。これにより、その遺伝的分化、多様性分布、隠蔽(亜)種の検出を行い、従来の形態に基づくアプローチよりも効率的な新種発見手法を確立します。
2. DNAバーコードライブラリーの構築:上記の解析で得られるDNA配列を蓄積し、日本近海の海洋生物との種内変異を網羅するDNAバーコードライブラリーを完成させ、オープンデータベースとして公開します。
3. 生物多様性の分布パターンの解明:汎太平洋地域における各国(ベトナム、タイ、マレーシア、シンガポール、インドネシア、フィリピン、韓国、オーストラリア、ニュージーランド)の新規環境DNAサンプルと、日本郵船との共同研究から得られる太平洋横断型の外洋の環境DNAサンプルを解析します。得られたデータから、海洋生物の組成変化を示す生物多様性ホットスポットや海洋生物多様性のグローバルなパターンを解明します。これらの調査は、ANEMONE調査プロトコルに基づき,環境DNA学会によって確立された標準化された方法を利用し、後世の研究との比較ができるように取り組みます。
本研究開発への参加・協力機関(五十音順):
石巻専修大学、かずさDNA研究所、熊本大学、島根大学、筑波大学、東京大学、東北大学、日本郵船、八戸工業大学、広島修道大学、北海道大学、和歌山高専、琉球大学
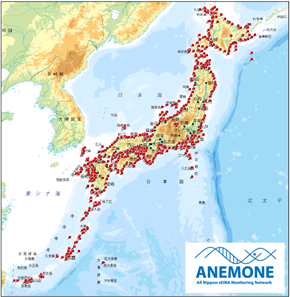
図1. ANEMONE データベースで観測がなされた測点

図2. 本プロジェクトで使用する日本郵船(右)と、実際に環境DNAを採取した点(左)
今後の展開
本プログラムから、以下の成果が期待されます。
1. 膨大な新種の発見、主要海洋生物のDNAバーコードライブラリーの確立:環境DNA調査は、ライブラリの不足から現行データが制限されています。本プロジェクトでは、データベースの内容は分類学者によって管理され、継続的に更新されます。このような信頼性の高い本データベースが本プロジェクトによって確立されることで環境DNA調査の対象種の拡大が見込め、今後の海洋生物多様性研究に不可欠な情報インフラとなることが期待されます。
2. 日本近海における生物多様性の形成過程の解明:本プロジェクトによって極めて高い精度で日本近海の海洋生物の種の分類を確立することができます。この基盤に基づき、広範な分類群に渡る海洋生物の、種内・種間の遺伝的多様性、分化、近親交配レベルを比較することで、遺伝的多様性の高い種を特定できます。このような遺伝情報と日本近海の地史情報を併せることで、各遺伝系統の多様化や、分散の歴史を通じた生物多様性の形成パターンを詳細に解明できます。
3. 未知の海洋生物多様性ホットスポットの発見: 日本および太平洋の環境DNAデータを通じて、隠蔽種、新種を含んだ各地域での種内・種間の遺伝的多様性の比較を、空前の規模で詳細に実施することで、これまで知られていなかった生物多様性のホットスポット(注4)を特定することが可能であると期待できます。さらにその生物多様性パターンの類似点と相違点を比較することで、海洋生物に普遍的な多様性の形成パターン要因の理解に大きく貢献すると期待されます。
1. 膨大な新種の発見、主要海洋生物のDNAバーコードライブラリーの確立:環境DNA調査は、ライブラリの不足から現行データが制限されています。本プロジェクトでは、データベースの内容は分類学者によって管理され、継続的に更新されます。このような信頼性の高い本データベースが本プロジェクトによって確立されることで環境DNA調査の対象種の拡大が見込め、今後の海洋生物多様性研究に不可欠な情報インフラとなることが期待されます。
2. 日本近海における生物多様性の形成過程の解明:本プロジェクトによって極めて高い精度で日本近海の海洋生物の種の分類を確立することができます。この基盤に基づき、広範な分類群に渡る海洋生物の、種内・種間の遺伝的多様性、分化、近親交配レベルを比較することで、遺伝的多様性の高い種を特定できます。このような遺伝情報と日本近海の地史情報を併せることで、各遺伝系統の多様化や、分散の歴史を通じた生物多様性の形成パターンを詳細に解明できます。
3. 未知の海洋生物多様性ホットスポットの発見: 日本および太平洋の環境DNAデータを通じて、隠蔽種、新種を含んだ各地域での種内・種間の遺伝的多様性の比較を、空前の規模で詳細に実施することで、これまで知られていなかった生物多様性のホットスポット(注4)を特定することが可能であると期待できます。さらにその生物多様性パターンの類似点と相違点を比較することで、海洋生物に普遍的な多様性の形成パターン要因の理解に大きく貢献すると期待されます。
用語説明
注1. 記載:生物の特徴を学術的な著作物上で発表すること。新種の命名や発表は、この記載の中で行われる。
注2. DNAバーコードライブラリー:生物の種の特徴を表すDNA配列のデータベース。
注3. 海綿動物:スポンジ状の体を持つ、最も原始的と考えられている動物群の名前。
注4. ホットスポット:生物の種数などが特に多い特定の地域。
注2. DNAバーコードライブラリー:生物の種の特徴を表すDNA配列のデータベース。
注3. 海綿動物:スポンジ状の体を持つ、最も原始的と考えられている動物群の名前。
注4. ホットスポット:生物の種数などが特に多い特定の地域。




